El Ministerio de Salud Pública y Bienestar Social (MSPyBS) del Paraguay ha anunciado que recibió los resultados preliminares del secuenciamiento de muestras paraguayas positivas para SARS-CoV-2, realizado en colaboración con el Brasil (1). Las autoridades del MSPyBS reportan dos genomas caracterizados como: la variante P.1 y la variante P.2, según la clasificación propuesta para estos virus (2).
Estos datos respaldan los resultados preliminares obtenidos en laboratorios de investigación en Paraguay (IICS/UNA; UNCA-CEDIC y Cyrlab), mediante otras técnicas moleculares que detectan la presencia de mutaciones características de estas variantes y que podrían estar mostrando la circulación de las mismas en nuestro país.
La variante P.1 es considerada una de las variantes de preocupación (VOC siglas del inglés variant of concern) debido a que presenta mutaciones en su genoma que están asociadas a cambios en algunas características biológicas del virus. Además, esta variante avanzó rápidamente en Brasil, dando duros golpes al país vecino (3).
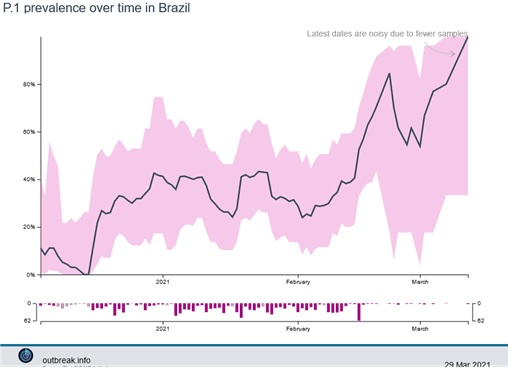
Manaus (Amazonas, Brasil) fue una de las ciudades más afectadas por la variante P.1 inicialmente, la cual fue detectada en la primera semana de diciembre de 2020 (4). Llamativamente, Manaus es una de las ciudades del vecino país con mayor tasa de casos previos de COVID-19 (5). A pesar de esto, los datos genómicos disponibles para la región son limitados.
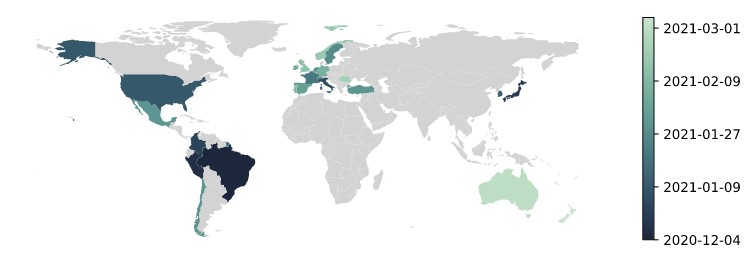
Ya a nivel mundial es sabido que una de las herramientas indispensables para la vigilancia genómica es el secuenciamiento del genoma viral. La secuenciación de virus de casos recientes arrojaría luz sobre la naturaleza y diversidad de los linajes que circulan en ciudades donde ocurre un aumento de casos, como ocurrió en Manaus.
Mutaciones en los virus y cuando preocuparse
Si bien la mayoría de las mutaciones pueden pasar desapercibidas (de hecho, en el genoma del SARS-CoV-2 ocurrieron miles desde que emergió en el 2019), un pequeño grupo de ellas podrían producir cambios de importancia biológica, como aumentar la transmisibilidad, la severidad o su habilidad de evadir la respuesta inmune.
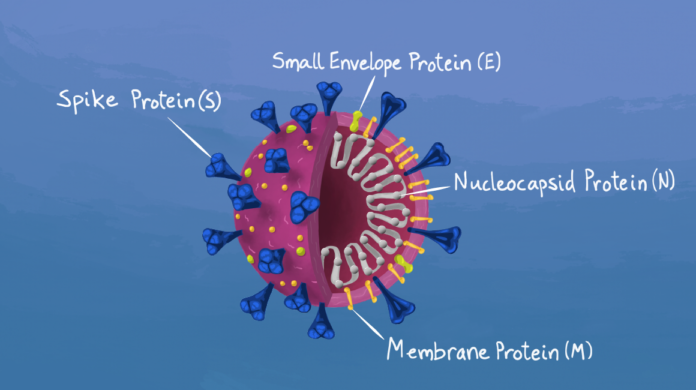
Una de las proteínas implicadas en los procesos mencionados es la proteína S (Spike) del virus, por lo que las mutaciones que ocurren en ella rápidamente llaman la atención de los investigadores.
Con el análisis de material genético y mediante el uso de herramientas bioinformáticas, se observa como las diferentes mutaciones (o cambios en el genoma del virus) se agrupan de acuerdo a sus características y se les asigna una serie de números y letras. Esas agrupaciones son denominadas “variantes”.
Así tenemos la variante B.1.1.7, que emergió en el Reino Unido en setiembre de 2020; la variante B.1.351, detectada en Sudáfrica en octubre de 2020 y la variante P1, detectada en Manaus, Brasil en diciembre de 2020; entre otras (4).
Estas tres variantes entran en la clasificación denominada “variante de preocupación” o VOC debido a que:
a) aumentan la transmisibilidad por lo que muestran un cambio perjudicial en la epidemiología;
b) producen un aumento de la virulencia o cambio en la presentación clínica de la enfermedad, o
c) producen una disminución de la eficacia de las medidas sociales y de salud pública, de los métodos de diagnóstico, vacunas y tratamientos disponibles. (6)
¿Qué sabemos de la variante P.1?
La variante P.1 también denominada 501Y.V3, fue detectada en Japón, en personas que habían viajado desde Manaus, Brasil. Posteriormente, tras algunas investigaciones, fue también confirmada la presencia de esa variante en la misma ciudad (Manaus). Recordemos que Manaus sufrió una intensa primera ola de COVID-19 que alcanzó su punto máximo en abril de 2020.
Luego de este duro golpe que sufrió la ciudad amazónica, se realizó un muestreo de donantes de sangre en octubre de 2020. Este estudio sugirió que el 76 % de la población tenía anticuerpos por lo que se presumía que al menos temporalmente, las personas serían inmunes.
Pero Manaus sufrió otra gran ola de la COVID-19 a inicios del año 2021, lo que llevó a científicos del Imperial College (Reino Unido) y la Universidad de Sao Paulo (Brasil) a estudiar el origen de la misma.
Descubrieron que la proporción de casos de COVID-19 causadas por la variante P.1, creció de 0 a 87 % en aproximadamente ocho semanas. Se encontró que P.1 era 1,4 a 2,2 veces más transmisible que otras variantes en Manaus, y se encontró que eludía entre el 25 % y el 61 % de la inmunidad protectora de una infección previa.
El plasma convaleciente de individuos infectados con variantes distintas a la 501Y.V3 (variante brasileña) neutralizaba la infección por la variante 501Y.V3 apenas con un sexto de la eficacia que tenía antes las demás variantes. Además, el plasma de pacientes vacunados con CoronaVac (una vacuna de virus inactivado) parece neutralizar los aislados (muestras) de 501Y.V3 con menos eficacia que a las de otras variantes.
Sin embargo, estas conclusiones deben ser tomadas con cautela, pues el tamaño de la muestra para este análisis fue pequeño)(7).
Sumado a esto, los datos más recientes sugieren un aumento en la severidad de los casos de COVID-19 causados por P.1 (8,9,10). La variante P.1 (Figura 3) contiene una serie de cambios o mutaciones (un total de 25 mutaciones, entre deleciones, inserción de nucleótidos y cambios de aminoácidos únicos).
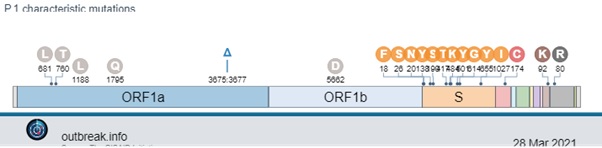
Tres de las mutaciones de P.1 que se asocian a cambios de importancia biológica ocurren en el dominio de unión al receptor (RBD) de la proteína spike: K417T, E484K y N501Y. La mutación N501Y está presente en las tres variantes (las detectadas en Brasil, Reino Unido y Sudáfrica). Esta mutación, así como la K417T, podrían estar asociada a la característica de las variantes de transmitirse con mayor facilidad, ya que aumentan la afinidad de la proteína del virus por su receptor celular.
La mutación E484K fue observada también en la variante de Sudáfrica y recientemente reportada en virus del linaje B.1.1.7 (11). Esta es la mutación que se cree da a las variantes cierta capacidad para escapar de las vacunas. Disminuyen su eficacia, aunque no las evaden totalmente.
Vigilancia basada en secuenciación
Expertos de todo el mundo coinciden en que, a pesar del inicio de la vacunación, queda un largo trecho por recorrer para el fin de la pandemia. En este momento, estamos atravesando un aumento de casos en casi todo el mundo.
Además de un esfuerzo por acelerar la vacunación y cerrar la brecha entre países ricos y pobres, continuar con las medidas de distanciamiento, testeo y aislamiento se han vuelto más importantes que nunca.
Sumado a todo esto, la vigilancia genómica toma un papel crucial, ya que la misma brinda información clave para ver el rumbo de la evolución de este patógeno. Y así utilizar esta información para el desarrollo de tratamientos, monitorear la eficacia de vacunas, de métodos diagnósticos y eventualmente tomar medidas de mitigación y control epidemiológico.
Referencias
- Nueva variante de SARS-CoV-2 circula en varios departamentos del país. Ministerio de Salud Pública y Bienestar Social. Marzo,2021.
- Rambaut, A., Holmes, E.C., O’Toole, Á. et al. A dynamic nomenclature proposal for SARS-CoV-2 lineages to assist genomic epidemiology. Nat Microbiol 5, 1403–1407 (2020). https://doi.org/10.1038/s41564-020-0770-5
- P.1 Lineage Report, Brasil. Outbreak Info.
- A standardized, open-source database of COVID-19 resources and epidemiology data. Outbreak Info.
- Faria Nuno, et al. (2021) Genomic characterisation of an emergent SARS-CoV-2 lineage in Manaus: preliminary findings. Virological.
- SARS-CoV-2 Variant Classifications and Definitions. (2021). CDC.
- De Souza et al., (2021). Variant: 20J/501Y.V3. CoVariants.
- Sabino E., et al 2021. Resurgence of COVID-19 in Manaus, Brazil, despite high seroprevalence. The Lancet.
- Challen R., et al. 2021. Increased hazard of mortality in cases compatible with SARS-CoV-2 variant of concern 202012/1 – a matched cohort study. medRxiv. doi.org/10.1101/2021.02.09.21250937.
- Coutinho R., et al. 2021. Model-based estimation of transmissibility and reinfection of SARS-CoV-2 P.1 variant. medRxiv org/10.1101/2021.03.03.21252706 (not per reviewed)
- Connecticut SARS-CoV-2 variant surveillance – Report 2021-03-25. Covid Tracker.
¿Qué te pareció este artículo?







 (16 votos, promedio: 4,63 de 5)
(16 votos, promedio: 4,63 de 5)














Excelente artículo de la Dra. Martinez. Mucha claridad para un tema complejo. Demuestra además que existe capacidad local para llevar adelante la vigilancia de las variantes y mutaciones del SarsCoV2. Felicitaciones!!
Quiero agregar a mi comentario anterior el agradecimiento a la Dra. Fabiola Roman quien también colaboró con este artículo