
Por Rhys Parry, Francisco J. Torres y Alberto A. Amarilla*
El coronavirus del tipo 2 causante del Síndrome Respiratorio Agudo Severo, abreviado SARS-CoV-2 o SRAS-CoV-2 (1), es un coronavirus responsable de la enfermedad por coronavirus de 2019 (COVID-19) (2, 3), cuya expansión mundial ha provocado la pandemia de la COVID-19, dejando más de 246 millones de personas infectadas y 4,9 millones de personas fallecidas (4).
Este coronavirus se descubrió por primera vez en Wuhan, China, en noviembre de 2019, siendo aislado por primera vez desde varios pacientes afectados por una neumonía atípica (2, 3).
SARS-CoV-2 posee un genoma de ARN de una sola cadena de polaridad positiva con un tamaño de casi 30.000 bases de pares nitrogenadas (2, 3). Este virus pertenece al género Betacoronavirus de la familia Coronaviridae, similar a otros coronaviruses patogénicos causantes de enfermedades respiratorias severas en humanos como el coronavirus causante del síndrome respiratorio agudo severo (SARS-CoV) y el coronavirus relacionado al síndrome respiratorio de Oriente Medio (MERS-CoV) (2, 3).
El medio de transmisión principal del SARS-CoV-2 es a través de microgotas producidas al respirar, hablar, estornudar o toser, pero también puede transmitirse a través de superficies contaminadas o alimentos congelados (5-7).
Los síntomas por infección de SARS-CoV-2 pueden conllevar desde una infección asintomática, pasando por síntomas leves parecidos a un resfriado, hasta provocar una enfermedad respiratoria aguda y neumonía severa en los seres humanos (8).
Los pasos iniciales de la infección por el SARS-CoV-2 implican la unión específica de la proteína S o espícula del coronavirus a su receptor celular ACE2 (enzima convertidora de angiotensina 2), que promueve la absorción viral y la fusión en la membrana viral con la membrana del huésped (9-10), tal como se encuentra ilustrada en la Figura 1.
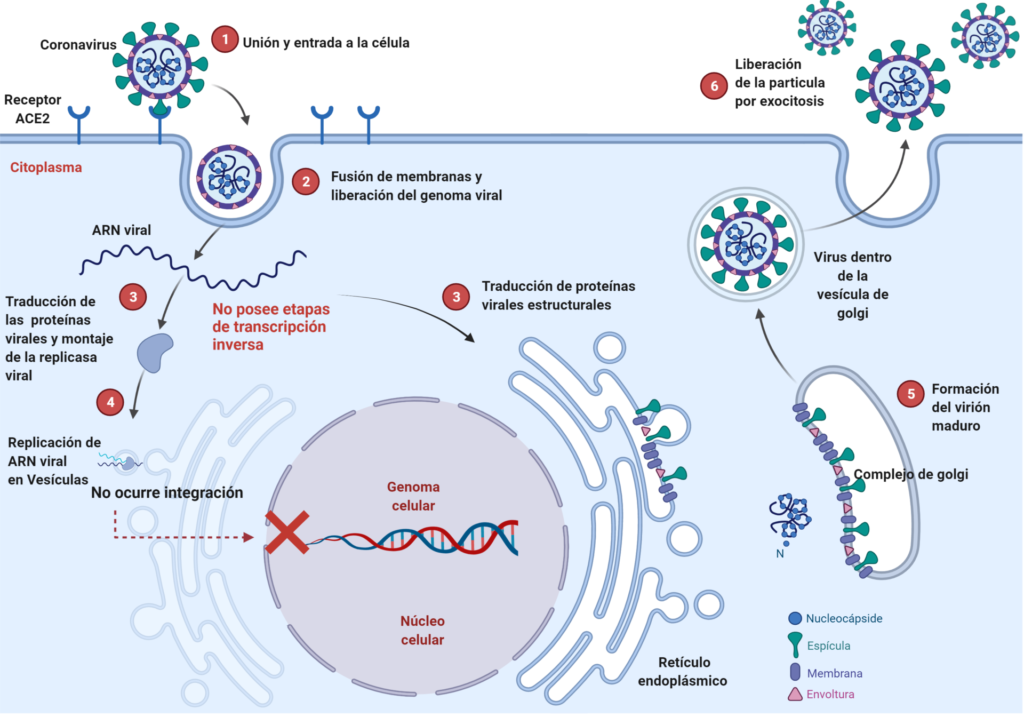
Después de la entrada, se produce la liberación del ARN genómico al citoplasma de la célula, iniciando la traducción inmediata de proteínas virales estructurales y no estructurales (Figura 1).
Muchas de las proteínas no estructurales están involucradas en la formación del complejo de replicación viral, un proceso que ocurre en vesículas de doble membrana asociadas al retículo endoplásmico. La formación de este complejo tiene como finalidad la producción de múltiples copias del genoma viral para su posterior empaquetamiento. Simultáneamente, las proteínas estructurales también son traducidas en las membranas del retículo endoplásmico y posteriormente direccionadas hacia las cisternas del Complejo Golgi, en donde la interacción con el ARN genómico recién producido y encapsulado con la proteína N da como resultado el montaje de nuevas progenies virales (Etapa 5, Figura 1).
Finalmente, los viriones son secretados de la célula infectada por de un proceso llamado de exocitosis (9-10). El genoma del SARS-CoV-2 no codifica ninguna enzima con capacidad de realizar la transcripción reversa del ARN viral a ADN viral, así como también carece de una enzima especializada para realizar la integración del genoma viral al ADN de la célula y, por lo tanto, no incorpora de forma natural copias del genoma viral dentro de las células huésped (2-3). Esta enzimas son también conocidas como “retrotranscriptasa e integrasa”.
Por otro lado, el ciclo de vida de los retroviruses tales como el virus de la immunodeficiencia adquirida (VIH), el causador del Sindrome de immunodeficiencia adquirida (SIDA), es bastante diferente al del SARS-CoV-2. El ciclo de replicación del VIH comienza cuando la partícula viral se fusiona a la superficie de la célula huésped mediante su proteína GP120 (Figura 2).
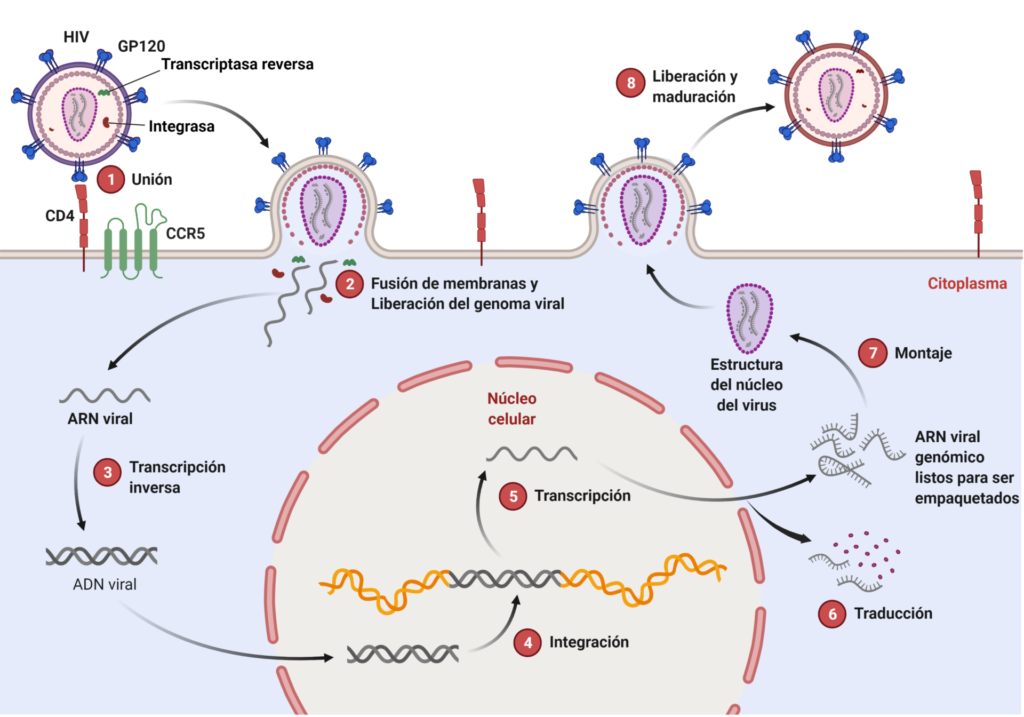
Luego, la cápside que contiene el genoma viral y las proteínas del virus ingresa a la célula. La capa de la cápside se desintegra y la proteína del VIH llamada retrotranscriptasa transcribe el ARN viral en ADN.
El ADN viral es transportado dentro del núcleo, donde la proteína viral llamada “integrasa” integra el ADN viral del VIH al genoma del huésped. La maquinaria de transcripción normal del huésped transcribe el ADN del VIH en múltiples copias del nuevo ARN del VIH. Parte de este ARN se convierte en el genoma de un nuevo virus, mientras que la célula usa otras copias del ARN para producir nuevas proteínas del VIH.
El nuevo ARN viral y las proteínas del VIH se mueven a la superficie de la célula, donde se forma un nuevo virion inmaduro, el cual sufre clivajes proteilíticos para crear un virus infeccioso maduro (11).
¿Es posible que el ARN viral del virus causador del COVID-19 pueda integrarse al genoma humano?
Ahora que entendemos algunas de las diferencias básicas más importantes entre el ciclo de vida de del SARS-CoV-2 y el VIH, pasaremos a discutir acerca de un reciente y controvertido artículo. El mismo fue publicado en la revista Proceedings of the National Academy of Sciences of the United States of America (PNAS) liderada por el Prof. Rudolf Jaenisch, investigador del departamento de biología del Instituto de Tecnología de Massachusetts (MIT) (12).
En este artículo, ellos demonstraron que la infección por SARS-CoV-2 puede culminar en la integración del ARN viral dentro del genoma de células humanas. Los autores sugieren que esta pueda ser la razón de un resultado positivo por prueba de RT-PCR en pacientes recuperados de la infección de la COVID-19.
Sin embargo, en una carta al editor de la PNAS liderada por un científico de la Universidad de Queensland, el Dr. Rhys Parry criticó la publicación original (13).
Parry y otros investigadores explicaron que la frecuencia de la supuesta integración del ARN viral al genoma humano elucidado por secuenciación de ácidos nucleicos de alto rendimiento -presentada por Jaenisch y sus colegas- ha sido extremadamente baja en ocurrencia (o abundancia), con lecturas identificadas reportadas entre 0,000003 a 0,0005 % del total. Por lo tanto, estos resultados podrían deberse simplemente a artefactos técnicos comúnmente asociados con la tecnología de secuenciación de alto rendimiento.
En un estudio dirigido por el grupo del Prof. Faulkne de la Universidad de Queensland no se reprodujeron los hallazgos liderados por el grupo del profesor Rudolf Jaenisch (14). Smits y sus colaboradores explicaron que la elección de la línea celular utilizada por el grupo de Jaenisch fue la células renales embrionarias humanas (HEK293T) que sobreexpresan el elemento retrotransponible llamado LINE1 (HEK293T-LINE1).
Estos elementos LINE1 (L1) están codificados en la mayoría de los de mamíferos y representa aproximadamente el 17 % del genoma humano (15-16) y su actividad ha sido relacionado a enfermedades crónicas como el cáncer (17-18). En los seres humanos, L1 transcribe un ARN mensajero bicistrónico que codifica dos proteínas chamadas ORF1p y ORF2p, esenciales para la movilidad de L1 (19). ORF1 codifica una proteína que tiene actividades de unión de ARN así como también puede actuar como una chaperona de ácidos nucleicos (20-24). El ORF2p posee actividades de endonucleasa (EN) y retrotranscriptasa (24-25).
Sin embargo, la sobreexpresión de LINE1 puede retrotranscribir (producir copias de ADN a partir de ARN) de cualquier ARN en el núcleo celular e integrarlo al genoma celular (26). En nuestro estudio, nosotros hemos utilizado células HEK293T que no sobreexpresan LINE1 y, curiosamente, no encontramos ninguna evidencia de integración del ARN del SARS-CoV-2 en el genoma del huésped (14).
Conclusión: nuestros resultados proveen sólidas evidencias de la no integración del ARN del SARS-CoV-2 en el genoma humano (13-14). Sin embargo, más estudios utilizando linajes celulares más relevantes y tejidos de personas infectadas necesitan ser conducidos para corroborar estas observaciones.
* Facultad de Química y Biociencias Moleculares, Universidad de Queensland, Australia
Autor correspondiente: Alberto A. Amarilla ([email protected])
Referencias
- World Health Organization WHO (2020). Naming the Coronavirus Disease (COVID-19) and the Virus that Causes it. World Health Organization (WHO) Technical guidance. Geneva: World Health Organization.
- Zhou P, Yang XL, Wang XG, Hu B, Zhang L, Zhang W, Si HR, Zhu Y, Li B, Huang CL, Chen HD, Chen J, Luo Y, Guo H, Jiang RD, Liu MQ, Chen Y, Shen XR, Wang X, Zheng XS, Zhao K, Chen QJ, Deng F, Liu LL, Yan B, Zhan FX, Wang YY, Xiao GF, Shi ZL. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature. 2020 Mar; 579(7798):270-273
- Wu F, Zhao S, Yu B, Chen YM, Wang W, Song ZG, Hu Y, Tao ZW, Tian JH, Pei YY, Yuan ML, Zhang YL, Dai FH, Liu Y, Wang QM, Zheng JJ, Xu L, Holmes EC, Zhang YZ. A new coronavirus associated with human respiratory disease in China. Nature. 2020 Mar; 579(7798):265-269
- John Hopkins University 2021. https://coronavirus.jhu.edu/map.html.
- Lydia Bourouiba. A Sneeze. N Engl J Med 2016; 375:e15
- Julian W. Tang, F.R.C.Path., and Gary S. Settles. Coughing and Aerosols. N Engl J Med 2008; 359:e19
- Matthew Meselson. Droplets and Aerosols in the Transmission of SARS-CoV-2. N Engl J Med 2020; 382:2063
- Wu Z, McGoogan JM. Characteristics of and Important Lessons From the Coronavirus Disease 2019 (COVID-19) Outbreak in China: Summary of a Report of 72 314 Cases From the Chinese Center for Disease Control and Prevention. JAMA. 2020 Apr 7; 323(13):1239-1242
- Megan Scudellari. How the coronavirus infects cells — and why Delta is so dangerous. Nature. 2021 Jul;595(7869):640-644
- Hu B, Guo H, Zhou P, Shi ZL. Characteristics of SARS-CoV-2 and COVID-19. Nat Rev Microbiol. 2021 Mar;19(3):141-154
- Eric O. Freed and Malcolm A. Martin. Human Immunodeficiency Viruses: Replication. Fields Virology, 6th Edition Edited by David M. Knipe and Peter M. Howley. Philadelphia, PA, USA. Lippincott Williams & Wilkins, 2013. 2456 pp
- Zhang et al., Reverse-transcribed SARS-CoV-2 RNA can integrate into the genome of cultured human cells and can be expressed in patient-derived tissues. Proc. Natl. Acad. Sci. U.S.A. 118, e2105968118 (2021)
- R. Parry, R. J. Gifford, S. Lytras, S. C. Ray, L. Coin, No evidence of SARS-CoV-2 reverse transcription and integration as the origin of chimeric transcripts in patient tissues. Proc. Natl. Acad. Sci. U.S.A. 118, doi:10.1073/pnas.2109066118 (2021)
- Nathan Smits, Jay Rasmussen, Gabriela O Bodea, Alberto A Amarilla, Patricia Gerdes, Francisco J Sanchez-Luque, Prabha Ajjikuttira, Naphak Modhiran, Benjamin Liang, Jamila Faivre, Ira W Deveson, Alexander A Khromykh, Daniel Watterson, Adam D Ewing, Geoffrey J Faulkner. No evidence of human genome integration of SARS-CoV-2 found by long-read DNA sequencing. Cell Reports, 36, (2021)
- Kazazian, H.H., Jr, and Moran, J.V. (2017). Mobile DNA in Health and Disease. N. Engl. J. Med. 377, 361–370
- E.S. Lander, L.M. Linton, B. Birren, C. Nusbaum, M.C. Zody, J. Baldwin, K. Devon, K. Dewar, M. Doyle, W. FitzHugh, et al., International Human Genome Sequencing Consortium. Initial sequencing and analysis of the human genome Nature, 409 (2001), pp. 860-921.
- JM, Li Y, Ju YS, Martincorena I, Cooke SL, Tojo M, et al. (ICGC Breast Cancer Group; ICGC Bone Cancer Group; ICGC Prostate Cancer Group) (August 2014). «Mobile DNA in cancer. Extensive transduction of nonrepetitive DNA mediated by L1 retrotransposition in cancer genomes». Science. 345 (6196): 1251343
- Ogino S, Nosho K, Kirkner GJ, Kawasaki T, Chan AT, Schernhammer ES, et al. (December 2008). «A cohort study of tumoral LINE-1 hypomethylation and prognosis in colon cancer». Journal of the National Cancer Institute. 100 (23): 1734–8
- Moran, J.V., DeBerardinis, R.J., and Kazazian, H.H., Jr (1999). Exon shuffling by L1 retrotransposition. Science 283, 1530–1534.
- H. Hohjoh, M.F. Singer. Cytoplasmic ribonucleoprotein complexes containing human LINE-1 protein and RNA. EMBO J., 15 (1996), pp. 630-639
- S.E. Holmes, M.F. Singer, G.D. Swergold. Studies on p40, the leucine zipper motif-containing protein encoded by the first open reading frame of an active human LINE-1 transposable element. J. Biol. Chem., 267 (1992), pp. 19765-19768
- E. Khazina, V. Truffault, R. Büttner, S. Schmidt, M. Coles, O. Weichenrieder. Trimeric structure and flexibility of the L1ORF1 protein in human L1 retrotransposition. Nat. Struct. Mol. Biol., 18 (2011), pp. 1006-1014
- S.L. Martin, F.D. Bushman. Nucleic acid chaperone activity of the ORF1 protein from the mouse LINE-1 retrotransposon. Mol. Cell. Biol., 21 (2001), pp. 467-475
- Q. Feng, J.V. Moran, H.H. Kazazian Jr., J.D. Boeke Human L1 retrotransposon encodes a conserved endonuclease required for retrotransposition. Cell, 87 (1996), pp. 905-91
- S.L. Mathias, A.F. Scott, H.H. Kazazian Jr., J.D. Boeke, A. Gabriel. Reverse transcriptase encoded by a human transposable element. Science, 254 (1991), pp. 1808-1810
- Doucet AJ, Wilusz JE, Miyoshi T, Liu Y, Moran JV. A 3′ Poly(A) Tract Is Required for LINE-1 Retrotransposition. Mol Cell. 2015 Dec 3;60(5):728-741
¿Qué te pareció este artículo?














